bitBiomeは、腸内細菌や皮膚常在菌などのヒト常在菌から生きた細菌のみをシーケンス解析する、「生菌選択的なシングルセルゲノムシーケンス(PMA-SAG-gel法)」(下記フロー図)を開発し、ヒト腸内細菌解析に用いた研究内容が公開されました。
今回開発したPMA-SAG-gel法では、細菌叢の生存プロファイルを株レベルで調べることが可能です。
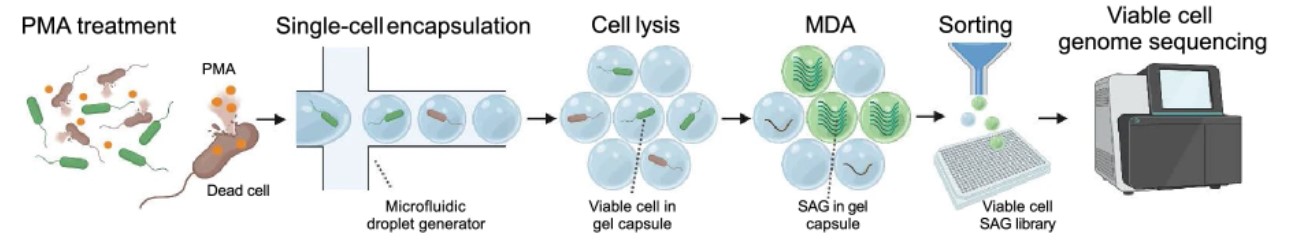
本研究ではPMA-SAG-gel法を用いることで、同じ細菌種でも株によって特定条件下での生存率が異なることがわかりました。このことは、生菌の情報を正確に得るためには株レベルでの評価を行うことが必要であることを示唆しています。
本手法により細菌叢中の生菌の特性評価、生菌製剤や糞便移植の品質評価に関する有益な情報が得られるため、細菌が人の健康に与える影響をより深く理解することにつながると期待しています。
本研究成果は、オープンアクセス科学誌『Scientific Reports』に2022年3月15日に掲載されました。
<掲載論文>
雑誌名:Scientific Reports
論文名:Strain-level profiling of viable microbial community by selective single-cell genome sequencing
主な著者: bitBiome㈱ CSO兼早稲田大学理工学術院 准教授 細川正人、bitBiome㈱ 研究開発部 リサーチャー 遠藤垂穂
掲載日:2022年3月15日
DOI: https://doi.org/10.1038/s41598-022-08401-y