早稲田大学とbitBiome株式会社の共同研究チームより、微生物シングルセルゲノム解析技術bit-MAP®で得られた遺伝子データのタンパク質発現により、標的とする未培養細菌の検出・分離を行った論文”Target enrichment of uncultured human oral bacteria with phage-derived molecules found by single-cell genomics“が公開されました。
本研究では、大規模に獲得した細菌のシングルセルゲノムデータから宿主特異的なファージ遺伝子を探索、タンパク質発現し、特異的プローブとして活用することで、標的細菌を検出することに成功しました。シングルセルゲノムデータを活用することで、未培養の有用微生物または病原性細菌の獲得など産業応用へつながることが期待されます。
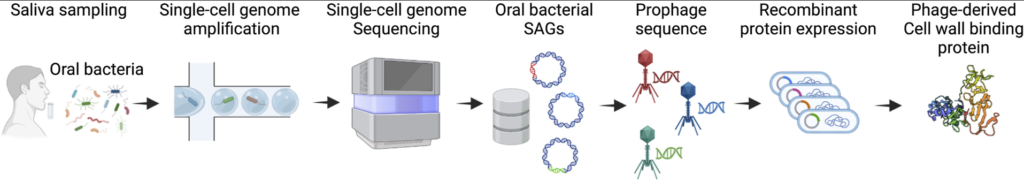
本研究成果は、科学誌『Journal of Bioscience and Bioengineering』に2023年5月13日に掲載されました。
bitBiomeは微生物シングルセルゲノミクス技術と世界最大級の微生物遺伝子データベースをコアに、最先端のインフォマティクスとロボティクスを活用した高効率の実験設備を掛け合わせることで、製薬、化学、食品業界でのバイオものづくりに貢献していきます。
<掲載論文>
雑誌名:Journal of Bioscience and Bioengineering
論文名:Target enrichment of uncultured human oral bacteria with phage-derived molecules found by single-cell genomics
主な著者(所属):細川正人(早稲田大学・bitBiome)、岩井直哉(早稲田大学)、佐伯達也(bitBiome)、有川 浩司(bitBiome)、依田 卓也(bitBiome)、津田宗一郎(bitBiome)、竹山春子(早稲田大学)(敬称略)
掲載日:2023年5月13日
DOI:https://doi.org/10.1016/j.jbiosc.2023.04.005