bitBiomeは、早稲田大学理工学術院の竹山 春子教授らとの共同研究にて、腸内細菌や皮膚常在菌などのヒト常在菌を単離培養することなく分析し、多様な細菌株のゲノムを正確かつ網羅的に獲得する「メタゲノム解析とシングルセル解析の統合データ解析法:シングルセルメタゲノミクス」を実現するためのフレームワーク「SMAGLinker」を開発しました。本研究成果は、Springer NatureグループのBioMed Central社が発刊するオープンアクセス科学誌『Microbiome』に2021年10月12日(火)に掲載されました。
SMAGLinkerは従来法よりも正確な遺伝子配列の割当が実行されており、腸内細菌・皮膚常在菌の解析において、完全性が高い高品質な細菌ゲノムを数多く出力しました(下図)。
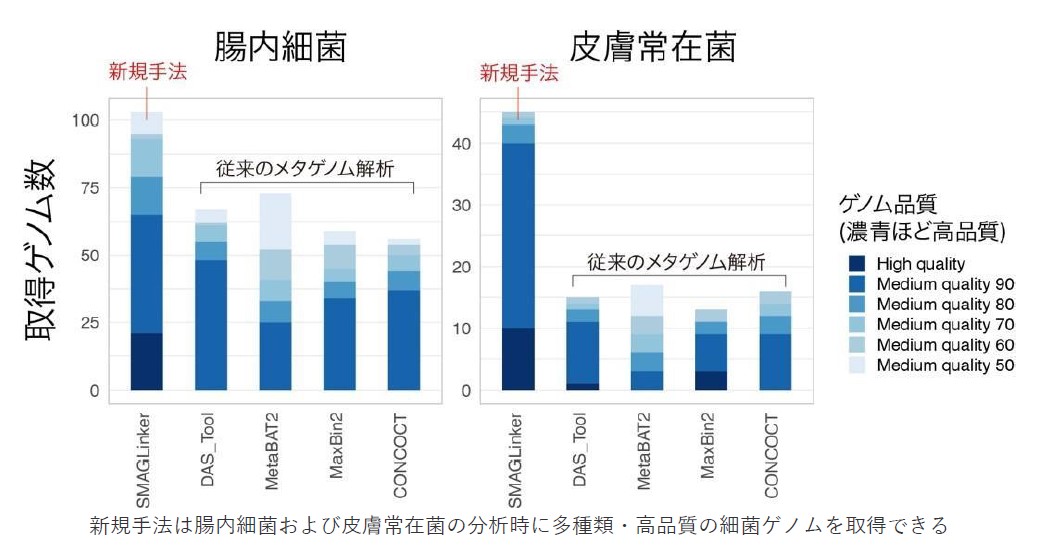
現在、ヒト常在菌の研究が世界中で活発に進んでいますが、同じ細菌種であっても外部からの遺伝子の取り込みや変異により異なる形質を持つことが明らかになっています。本研究で開発したSMAGLinkerにより、ヒト常在菌ゲノムが正確に決定されることで、個々人によって異なる細菌株の特性を正しく理解し、ヒトとの相互作用による健康や疾患との関わりについての理解が進むことが期待されます。
SMAGLinkerによるバイオインフォマティクス解析もパートナー様に提供しておりますので、ご関心がございましたら、info@bitbiome.co.jpまでお気軽にお問い合わせ下さい。
<掲載論文>
雑誌名:Microbiome
論文名:Recovery of strain-resolved genomes from human microbiome through an integration framework of single-cell genomics and metagenomics
主な著者: bitBiome㈱ CSO兼早稲田大学理工学術院 准教授 細川正人、bitBiome㈱研究開発部 ジェネラルマネジャー 有川浩司
掲載日(日本時間):2021年10月12日(火)
DOI:https://doi.org/10.1186/s40168-021-01152-4
<早稲田大学からのプレスリリース>
細菌叢から細菌ゲノムを個別取得するデータ解析フレームワーク「SMAGLinker」を開発
~シングルセルとメタゲノムの統合による高精度ゲノムデータの実現~
【bitBiomeについて】
bitBiomeは、地球上のあらゆる微生物の設計図=ゲノムデータを最高解像度で蓄積し、あらゆる産業をupdateします。独自のシングルセルゲノム解析技術bit-MAP®により、これまで培養・解析が極めて難しかった微生物から高効率で全ゲノム配列を取得し、質・量ともに他を凌駕する微生物遺伝子データベースを構築します。これを起点とし、産業パートナーと協調してデータからモノを創り出す革新的バイオ生産プラットフォームを展開します。詳細については https://www.bitbiome.co.jp/をご覧ください。
お問合せ先:info@bitbiome.co.jp