bitBiomeと早稲田大学の研究チームより、論文”A single amplified genome catalog reveals the dynamics of mobilome and resistome in the human microbiome”が公開されました。
本研究ではがん・炎症性腸疾患などの患者と健常者を含む日本人被検者 51 名から、世界最大規模3万個の口腔内細菌および腸内細菌のシングルセルゲノム解析を行いました。
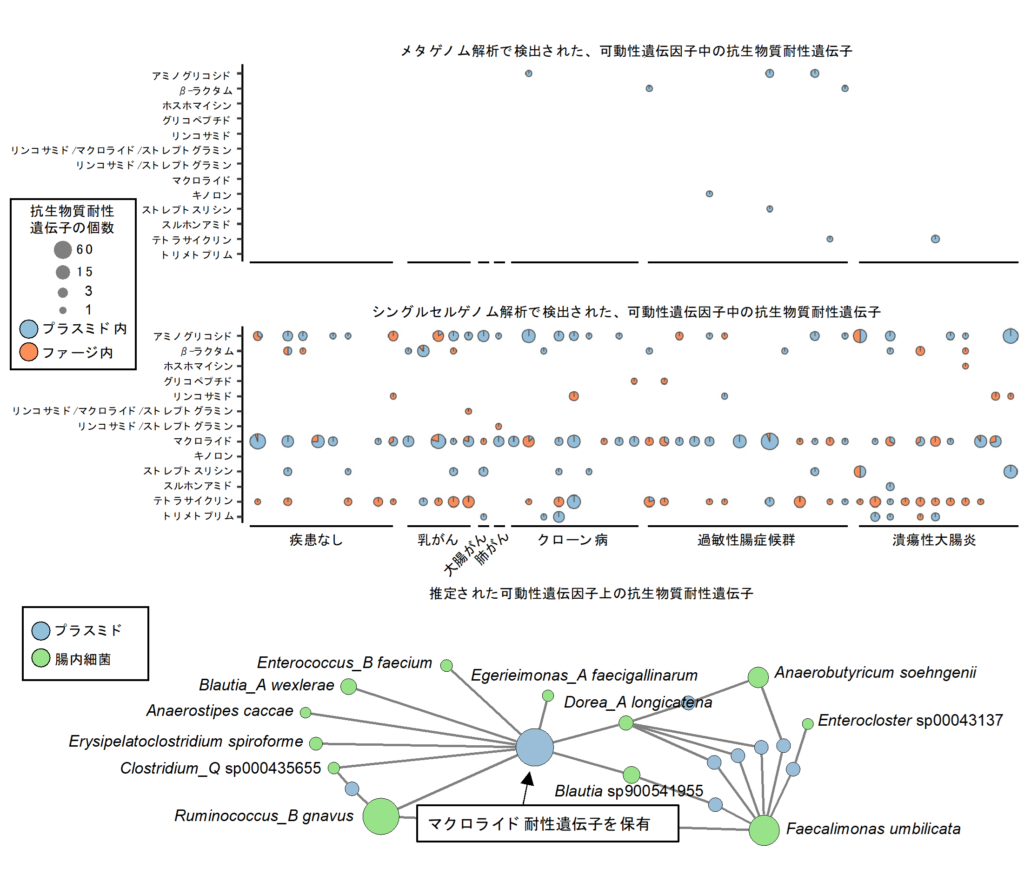
その結果、従来法では見落とされていた数百種の細菌のゲノム・遺伝子を発見しました。また、抗生物質耐性遺伝子やその「運び屋」の存在が個々の細菌単位で明らかになり、細菌間での遺伝子のやり取りを詳細に調査することが可能になりました。
微生物シングルセルゲノム解析bit-MAP®は、細菌の抗生物質耐性の広がり方をより深く理解する手がかりを提供し、常在菌を対象とした個別化医療や新たな抗生物質耐性対策の開発に貢献していく事が期待されます。
早稲田大学からのプレスリリース『ヒト常在菌の個別解析、新時代へ〜3万個の細菌ゲノム解読、抗生物質耐性遺伝子を追跡』にて詳細をご紹介していますので、併せてご確認ください。
<掲載論文>
雑誌名:Microbiome
論文名:A single amplified genome catalog reveals the dynamics of mobilome and resistome in the human microbiome
著者名:Tetsuro Kawano-Sugaya1† , Koji Arikawa1,2†, Tatsuya Saeki1, Taruho Endoh1, Kazuma Kamata1, Ayumi Matsuhashi1 and Masahito Hosokawa1,2,3,4,5*
1. bitBiome 株式会社
2. 早稲田大学 先進理工学研究科
3. 産総研・早大 生体システムビッグデータ解析オープンイノベーションラボラトリ
4. 早稲田大学 ナノライフ創新研究機構
5. 早稲田大学 先進生命動態研究所
掲載日時(現地時間):2024 年 10 月 2 日
DOI:https://doi.org/10.1186/s40168-024-01903-z
■bitBiome株式会社 概要
bitBiome株式会社は、世界最大規模の微生物ゲノムデータベース bit-GEMを作り上げ、バイオものづくりに貢献する酵素探索・改変プラットフォームbit-QEDを展開しています。bit-GEMには、従来は解析できなかった微生物の遺伝子情報を正確に読める世界初の「微生物シングルセルゲノミクス技術bit-MAP®」が活用されています。bitBiomeは微生物シングルセルゲノミクス技術と世界最大の微生物ゲノムデータベースをテコに、脱炭素や循環型社会、医療イノベーションへの貢献を通じたバイオエコノミーの実現に貢献していきます。詳細については、https://bitbiome.co.jp/をご覧ください。